二聚超氧化物歧化酶的结构性变换和肌萎缩性侧索硬化症
- 2012-11-06
- 专题
1.肌萎缩侧索硬化症(ALS) 和突变型超氧化物歧化酶(SOD)
肌萎缩侧索硬化症(ALS)是一种渐进的麻痹性疾病,其特点是上下运动神经元选择性退化。1,2)虽然ALS是一个显著的偶发性疾病,但是~10%的案例是常染色体显性遗传(家族性ALS(fALS))和部分fALS案例是由SOD1基因突变导致的。3,4)SOD1产品基因,细胞质Cu,Zn-超氧化物歧化酶(SOD1),是一种广泛表达酶,能够催化超氧自由基歧化反应。5)
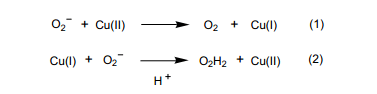
SOD(Cu/Zn)的晶体结构已经确认,8) Fig.1 (PDB, 1spd_x)所示它的二聚体结构。铜和锌通过组氨酸咪唑环阴离子的连接在一起。
一些数据表明:SOD1突变的结果除了功能缺失导致肌萎缩侧索硬化症(ALS)以外,还有另外的结果。例如,一些fALS-相关的突变体SOD1s保留了完整的酶活性。6)此外,SOD1能够导致小鼠失去知觉,不显示ALS症状,然而转基因小鼠表现出fALS-相关突变体G93A SOD1延伸性ALS-类似症状尽管也有内源性小鼠SOD1表现。7)最终在这个转基因小鼠试验中过表达人类野生型SOD1的失败用于缓解ALS症状。7)
一种假设解释说SOD1功能性的收获是突变体的错折叠改变了其催化机制,从而允许了过氧亚硝酸盐9)和有可能的过氧化氢生成。10)另外一个重要的假说认为这个毒性源于SOD1细胞内聚集体。SOD1包涵体,与anti-抗体反应,是ALS 患者常见的运动神经元和附近的星形胶质细胞供给体。11)
然而,当我们考虑到蛋白质氧化钙性有助于聚集体和蛋白酶抵抗力的时候,这两种假设也不是相互排斥的。蛋白质聚集是很多种神经性失常变性疾病的病理性特征,12)包括Huntington’s,Alzheimer’s和 Parkinson’s等疾病。每一种情况下,SOD1s 突变体错折叠和聚合倾向的可能性原理通过超过100不同的突变导致一个常见的表现型。尽管SOD1聚集体可能有的内在毒性或者是通过螯合分子伴侣导致的运动神经元毒性阻碍了蛋白酶的正常运作,但是SOD1聚集体的毒性起源尚未明确。
要了解ALS的发病机制,我们必须弄清楚如何改变SOD分子能诱导分子的损伤。为了进行这样的调查,我们已经开始弄清楚了功能增益的原因和SOD聚集体在溶液中的机制。
SOD1的反应机制已经被许多学者进行了研究。最近Nishida等人对于这种酶基于通过使用模型化合物所得的结果有了一个新的假设机制。13)在上述第二步骤中 (2)我们已经指出了Cu--OOH形式的中间体形成的重要性(见图表-I),这种过氧化氢能够从野生型酶中立即祛除,因为过氧化氢和copper(II)离子与周围的有机基团分子间作用基本上可以忽略不计。

Siddique等人7)已经确定了人类SOD的晶体结构,还有另外两种SOD结构,并且已经确定fALS突变不能改变任何活性点的残基包括基质中的静电识别,金属离子的结扎或者是活性点通道的形成,但是能够检测到铜离子(Ⅱ)周围细微的变化。基于准确研究典型结构和蛋白质中疏水材料的积极影响的基础上,6个fALS突变体有可能破坏亚单位折叠或者二聚体结构。但是,我并不认为这些对亚单位折叠或者二聚体结构的破坏性影响可以足够的解释所有fALS 发病原因。
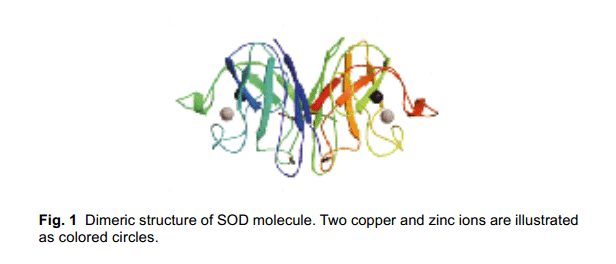
1997年, Yim等人报道了一个fALS 突变体(Gly93Ala=G93A)表现增强自由基生成活性的作用, 而它的歧化活性与野生型酶一样。14)图2为含有H2O2和人SOD 酶溶液中DMPO-OH radical加成物生成物ESR 波谱。据他们报道,突变体中自由基生成的活性,在低浓度H2O2条件下通过自旋捕捉法,相对于wild-type和G93A活性是增强的,wild-type<G93A<A4V,但是上述观测到的原因还没有详尽的描述。
2. “增益功能”SOD酶起源与Cu(II)-过氧化氢加合物独特的反应性能
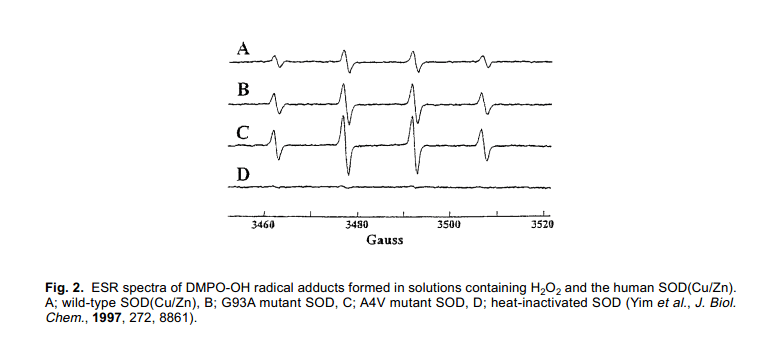
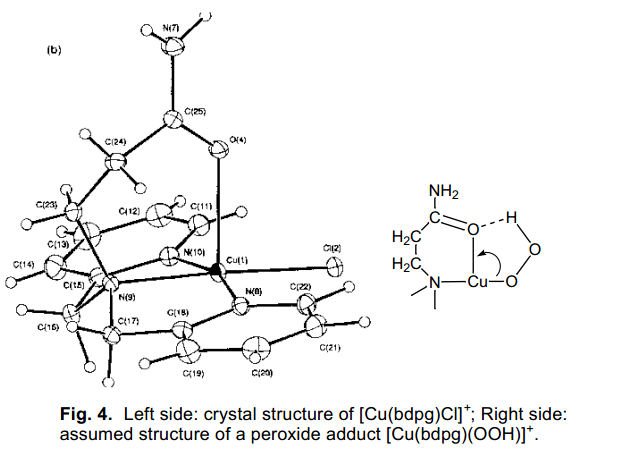
为了得到SOD突变体结构变化和fALS发病机制之间相关的一个全面的解决方案,我们研究了Cu(II)-OOH的反应性能,用来作为一个SOD反应中的重要中间体。为了这个目的,我们已经合成了很多种Cu(II)化合物,这种化合物都含有N,N-双(2-吡啶甲基)胺配体,如图3所示。15)所有的Cu(II)化合物结构特点都十分相似(作为例子, [Cu(bdpg)Cl]+晶体结构如图4所示)。在过氧化氢的参与下,预期生成了加成产物如图4右边所示,这个结果与用环己烷等反应一致,并且我们发现Cu(II)化合物过氧化物加合物的反应性能高度依赖于配体系统中的R,这是因为系统中不同的导致Cu(II)离子周围细微的结构变化的结果。
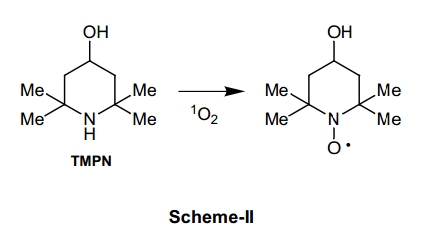
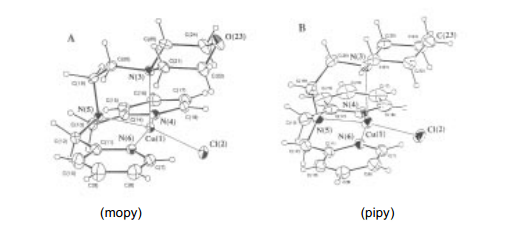
我们已经测定了含有Cu(II)络合物和自旋捕捉试剂溶液的ESR波谱,例如PBN(α-苯基-N-叔丁基氮氧化物)和TMPN (N,N,N',N'-四甲基-4-哌啶醇), 分别用于OH自由基和单重氮(1∆g)(图表-II)的特定试剂。16)当Cu(II) (tpa) (=三(2-吡啶甲基)胺)或者(bdpg)络合物与H2O2和PBN混合时,由于PBN自由基的生成,我们检测不到ESR信号。然而,溶液中含有Cu(tpa)络合物的情况下,由于TMPN (图表-II )相应的硝酸自由基的生成,我们能够检测到强峰,含有Cu(bdpg)络合物的溶液则没有这种情况。尤其是在做Cu(pipy)Cl+和Cu(mopy)Cl+之间的比较时非常有趣。15)这两种化合物的结构特点非常相似,除了Cu(mopy)Cl+络合物morphorin 环上的氧原子被替换成了Cu(pipy)Cl+络合物上面的-CH2的差别(见下图)。
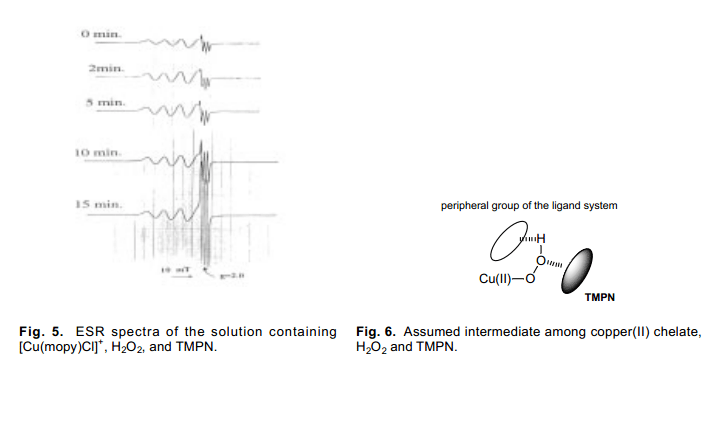
Cu(pipy)Cl+过氧化氢参与条件下, 我们观测不到硝酮自由基的形成。与此相反,由Cu(mopy)Cl+ 络合物生成的自由基能够检测到有很高的活性,如图5。在Cu(II)[Cu(Hphpy)Cl]+络合物里面我们也观测到了类似的高活性的TMPN 自由基的生成。这种情况下,类似的Cu(mopy)Cl+ 络合物, 在Cu(II)溶液中加入H2O2,由于铜离子的原因,诱导不了ESR spectrum发生变化;但是添加TMPN 会导致ESR 信号属性发生急剧的变化( i.e. , 由于铜原子发生超精细结构变化)。上述这些所有的假设是建立在当三种试剂存在于溶液中的时候Cu(II)络合物、过氧化氢、TMPN的发生(见图6), 当溶液中中间物形成时可以观测到过氧化氢独特的反应性能。
以上事实表明Cu(II)-OOH的反应性能是由中间体的结构性质所决定的(见图6)i.e. , 通过Cu(II)-OOH,外围基团和基材之间的相互作用而得。13)在这里应当指出虽然过氧化氢被认为相对惰性且对细胞无毒,但是我们目前的结果明确的表明某些Cu(II)螯合物可以激活过氧化氢,表现出类似于单重氮(1∆g)的高活性。
为了进一步获得Cu(II)-OOH物种反应性能,我们已经测得了Cu(II)化合物和过氧化氢溶液的ESI-质谱。当Cu(Me-bdpg)Cl溶液中加入过氧化氢(见图3),由ESI-质谱测得生成了[Cu(bdpg)Cl],而不是[Cu(dpal)]。17)这些都清楚的表明Cu(II)-OOH物种能够切割氧化缩氨酸C-N键,而不是水解,因为因为水解断裂就可能从Cu(Me-bdpg)化合物中得到Cu(dpal)种。
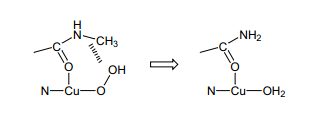
我们还发现某些Cu(II)络合物在过氧化氢的存在下,蛋白质发生分解,在淀粉样蛋白beta-肽(1-40)蛋氨酸残基的氧合硫原子18)处显示出很高的活性。19)所有的事实可能表明突变型SOD 的“增益功能”是由于在SOD 反应进程中有一个长久的高活性的Cu(II)-OOH作为中间体形成。突变型SODCu(II)周围的结构发生很大的变化,这给Cu(II)-OOH的反应能行了一个意想不到的影响,正如我们文件所观测到的。通过Cu(II)-OOH突变型SOD的C-N 键的断裂使得SOD表面发生很大的变化,形成SOD 酶不稳定的二聚体。20)因此,具有高反应性Cu(II)-OOH 基团的形成和存在很有可能是fALS 发病机制中氧化应激的一个内在起源,这与我们最近的含有SOD酶的不稳定性二聚体的研究有一定的关联。21,22)
3二聚体SOD分子分解成单体
如前文所述,蛋白质聚集被广泛的认为是很多种神经系统疾病一个常见的病理性特征,包括Huntington’s, Alzheimer’s, and Parkinson’s 等疾病,SOD1聚集体本身的毒性或者是由螯合分子伴侣阻碍蛋白酶体正常的功能运作所产生的运动神经元毒性所致。
2004年, Rakhit 等人报道了SOD1通常是一个二聚体酶,即单体解离之前野生型和突变体蛋白的集合体。23)他们使用“Dynamic Light Scattering (DLS)”方法了检测二聚体SOD 的单体解离。最近我们也曾报道了毛细管电泳法(CE)是非常适合进行调查研究蛋白质构象变化和蛋白质在溶液中的聚集状态的一个方法。24)举一个例子,图6显示两个SOD 和运铁蛋白的CE曲线图。25)尽管这两种酶的浓度时相同的,但是他们的峰的强度彼此有很大的不同,这个已经被合理化的事实是因为溶液中SOD 具有一个刚性二聚体结构,而apo-运铁蛋白二聚体结构更为灵活。26)

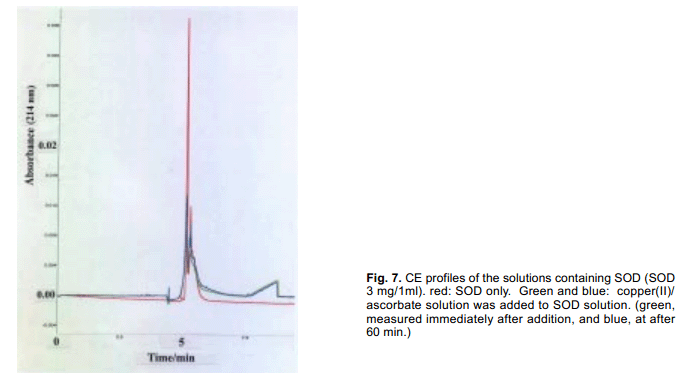
如图7所示当Cu(II)/抗坏血酸溶液中加入SOD分子的时候,由于二聚体SOD的生成,我们可以观测到峰值强度急剧下降;我们的实验系统和Rakhit 等人报道是一样的。这就清楚的表明二聚体SOD分子的解离可以通过CE法很容易的检测到。
我们也已经发现过量的过氧化氢导致SOD分子二聚体结构出现松动或者是解离。26)过氧化氢的参与是二聚体SOD 解离的起源是很明确的,26) Kakhit等人很有可能在系统中使用的氧化剂为过氧化氢,偶发性ALS很可能与过氧化氢的存在有关,同样的讨论也可以应用于偶发性朊病毒病的阐述。
通过使用抗体的方法来迅速的纯化SOD1,并且用质朴分析法进行耦合连接,Sato等人已经在29 SOD1-突变fALS 患者身上检测出了 其红细胞野生型和突变型SOD1相对积累水平。22)他们观测到检测不到SOD1突变体的患者持续疾病时间最短。尽管疾病发病年龄与SOD1突变体的数量没有直接的关系,但是令人信服的是它显示了疾病时间与突变体积累的一个很强的逆相关系。换句话来说,不太稳定的突变体被发现可能加速病情进展。这一令人惊奇的发现意味着尽管错误折叠的SOD1突变体的不稳定形式加剧了疾病潜在毒性的进展,但是SOD1 突变体稳定性与疾病发病没有一一对应的关系。因此SOD1二聚体分子分裂成为错误折叠单体应该会是APS 发病机制过程中必不可少的重要步骤。很明显过氧化氢在SOD1单体26)形成的过程中起了一个很重要的作用,所以我们应该注意到人体中过量的过氧化氢的生成,特别是二聚体(III)与谷胱甘肽环和其他相关的系统。27)
4.偶发性朊病毒病Cu(II)-OOH
在英国,1980年到大约1996年之间,大约有750,000 牛感染了BSE (牛海绵状脑病,传染性海绵状脑病之一)的牛被屠宰供人类食用,目前人们公认为TSEs 中心部分是将正常的细胞朊蛋白(PrPC)翻译转化为一个非正常的的亚型痒病PrP (PrPSc),这种疾病具有一个高-β-折叠片段内容并且与传染性疾病有关。28)人们普遍的认为PrPC是一种含铜蛋白 (在N-末端的非结构化区域至多有4个铜离子具有八面体重复部位)。小鼠和鸡PrPC的重组体的研究分析随着PrPC铜络合物的形成使我们得到了一个“增益功能”的重要发现;PrPC 已经被证明有助于直接影响细胞SOD 活性的功能。
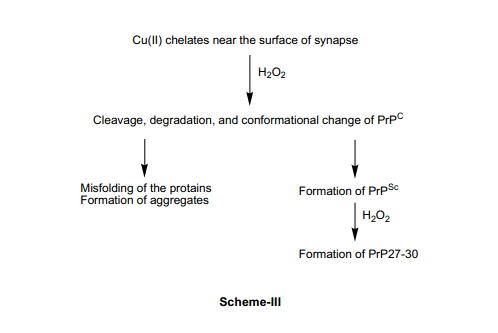
错误折叠朊蛋白 (PrPSc) 最终杀死充满大脑中类似于海绵一样的神经元和叶状物。除了PrPSc以外, 还有另外一个从受影响的脑中萃取出来的耐蛋白酶PrP(27-30 kDa), 被叫做PrP27-30。在这里应该注意的是PrP27-30 唯一来自于PrPSc(而不是PrPC), 这个和PrPC至PrPSc直接已经确定顺序的氨基酸没有差别。基于这些事实,我们可以假定,PrPSc铜离子周围的化学环境应该和PrPC是有所不同的;这种情况和野生型与突变型SOD酶铜离子周围出现的差异性相似。因此,PrPSc的“增益功能”很有可能归因于“高活性”Cu(II)-OOH 结构可能发生如描述中的突变体SOD分子,导致在铜离子周围的肽键发生断裂(大约在90 site处), 然后得到危险的PrP27-30;后面的蛋白质就可能比较类似错误折叠SOD单体。除了这一点儿以外,PrPC和PrPSc 中的铜离子很有可能与过氧化氢反应得到Cu(II)-OOH类产物,这很有可能严重影响到PrPC 譬如甲硫氨酸残基的氧合,构象的变化(即PrPSc的形成), 以及过氧化氢参与下蛋白质的降解(见图表-III)。天然朊蛋白29-32)中观测到的几个实验事实与我们的讨论是一致的。所有的这些研究结果都支持着我们的提案,来源于PrPSc SOD功能结构和代谢异常的铁离子27)的过氧化氢很有可能是零星朊病毒疾病氧化应激力的真正来源。
参考文献
[1] R. G. Smith and S. H. Appel, Annu. Rev. Med. , 1995, 46,133.
[2] R. H. Brown, Jr., Cell , 1995, 80, 687.
[3] H.-X. Deng, et al., Science, 1993, 261 , 1047.
[4] M. W.-Pazos, et al., Science, 1996, 271 , 515.
[5] I. Fridovich, Annu. Rev. Biochem., 1995, 64, 97.
[6] D. R. Borchelt, et al., Proc. Natl. Acad. Sci. USA., 1994, 91,8292-8296.
[7] L. I. Brujin et al., Science, 1998, 281 , 1851-1854.
[8] J. A. Tainer, E. D. Getzoff, J. S. Richardson, and D. C.Richardson, Nature, 1983, 306 , 284.
[9] A. G. Estevez et al., Science, 1999, 286 , 2498-2500.
[10] M. B. Yim et al., Proc. Natl. Acad. Sci. USA., 1996, 93, 5709-5714.
[11] M. Watanabe et al., Neurobiol. Dis., 2001, 8 , 933-941.
[12] B. Caughey and P. T. Lansbury, Annu. Rev. Neurosci., 2003,26, 267-298.
[13] Y. Nishida and S. Nishino, Z. Naturforsch. , 2001, 56c , 144-153; Y. Nishida, Med. Hypothesis Res., 2004, 1 , 227.
[14] M. B. Yim et al., J. Biol. Chem., 1997, 272 , 8861.
[15] T. Kobayashi, T. Okuno, T. Suzuki, M. Kunita, S. Ohba, and Y. Nishida, Polyhedron, 1998, 17, 1553.
[16] S. Nishino, T. Kobayashi, M. Kunita, S. Ito, and Y. Nishida, Z. Naturforsch., 1999, 54c , 94.
[17] S. Nishino, M. Kunita, Y. Kani, S. Ohba, H. Matsushima, T.Tokii, and Y. Nishida, Inorg. Chem. Communications , 1999,3 , 145.
[18] S. Nishino and Y. Nishida, Inorg. Chem. Communications, 2001, 4, 86; Synth. Reac. Inorg. Metal-Org. Nano-Metal Chem. , 2005, 35, 677.
[19] S. Nishino, A. Kishita, and Y. Nishida, Z. Naturforsch. J. Biosciences, 2001, 56c , 1144.
[20] P. Cioni, A. Pesce, B. Morozzo, S. Castelli, M. Falconi, L. Parrilli, M. Bolognesi, G. Strambini, and A. Desideri, J. Mol.Biol. , 2003, 326 , 1351.
[21] O. Matsumoto and Fridovich, Proc. Natl. Acad. Sci. USA. ,2002, 99, 9010.
[22] K. Yamanaka and D. W. Cleveland, Neurology , 2005, 65,1859.
[23] R. Rakhit et al., J. Biol. Chem., 2004, 279 , 15499-1550.
[24] A. Kishita and Y. Nishida, Annu. Report CIN , 2004, 1; Nishida et al., Synth. Reac. Inorg. Metal-Org. Nano-Metal Chem., 2005, 35, 379; Nishida et al., Z. Naturforsch., 2007, 62b , 205.
[25] Y. Sutoh et al., Chem. Lett., 2005, 34, 141.
[26] Y. Nishida et al., Z. Naturforsch. , 2006, 61c , 273.
[27] Y. Nishida, et al., Z. Naturforsch. , 2007, 62c , 608; Y. Nishida, Recent Res. Devel. Pure & Appl. Chem. , 1999, 3 , 103; Med. Hypothesis Res., 2004, 1 , 227.
[28] B. Caughey, Trends Biochemical Sciences, 2001, 25, 235.
[29] J. R. Requena, N. D. Dimitrova, G. Legname, S. Teijira, S. B. Prusiner, and R. L. Levine, Arch. Biochem. Biophys., 2004, 432 , 188.
[30] H. E. M. MaMahon, A. Mange, N. Nishida, C. Creminon, D. Casanova and S. Lehmann, J. Biol. Chem., 2001, 276 , 2286.
[31] R. Requena, D. Groth, G. Legname, E. R. Sradtman, S. B. Prusiner, and R. L. Revine, Proc. Natl. Acad. Sci. USA., 2001,98, 7170.
[32] N. T. Watt, D. R. Taylor, A. Gillott, D. A. Thomas, W. S. Perera, and N. M. Hooper, J. Biol. Chem. , 2005, 280 , 35914; B. J. Tabler, S. Turnbull, N. J. Fullwood, M. German, and D. Allsop,Biochem. Soc. Trans., 2005, 33, 548; B. J. Tabler, O. M. A.E.-Agnaf, S. Turnbull, M. J. German, K. E. Paleologou, Y. Hayashi, L. J. Kooper, N. J. Fullwood, and D. Allsop, J. Biol. Chem., 2005, 280 , 35789; N. T. Watt and N. M. Hopper,Biochem. Soc. Trans., 2005, 33, 1123; S. Fernaeus, K. Reis,K. Bedecs, and T. Land, Neuroscience Lett., 2005, 389 , 133.



 沪公网安备 31010602001115号
沪公网安备 31010602001115号